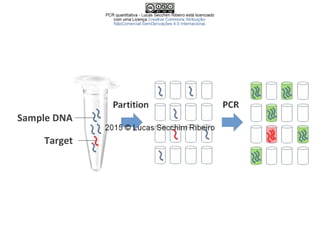
PCR Quantitativa - Lucas Secchim Ribeiro (PGMicro UFMG 2015-1)
- 1. Programa de Pós-Graduação em Microbiologia Biologia Molecular de Micro-organismos (MIC842) ICB/UFMG PCR quantitativa Teoria e Aplicações Lucas Secchim Ribeiro Laboratório de Interação Microrganismo-Hospedeiro
- 2. • PCR Histórico Teoria Aplicações • Desenho de primers • qPCR Metodologias Cálculos Aplicações PCR Digital
- 3. Dogma central da Biologia Molecular • Francis Crick / 1956
- 6. PCRPCR
- 7. PCR - Polymerase Chain Reaction
- 8. Funções da PCR • Distinguir uma sequência-alvo específica de uma grande quantidade de background; • Amplificação de cópias de uma sequência específica a partir de pequenas quantidades do DNA modelo.
- 9. Aplicações da PCR “convencional” • Diagnóstico • Avaliação de mutações • Detecção de patógenos (bactérias, fungos, vírus, protozoários) • Tipagem para transplantes - MHC • Testes forenses • Paternidade • Investigações criminais • Sequenciamento e clonagem • Epidemiologia molecular e bioinformática • Análise de OGM • ...
- 10. PCR “convencional” Reagentes básicos Iniciadores senso/antissenso (primers forward/reverse) Cátions (Na+ /K+ e Mg2+ ) DNA polimerase com DNA modelo Deoxinucleotídeos (dATP, dCTP, dGTP, dTTP)
- 11. Importância do tampão e íon potássio na PCR • pH 8,3 pH 7,2 a 72º C • Tris/ Tris-Cl •Função enzimática da DNA polimerase • K+ • Reduz a repulsão entre DNA/DNA e DNA/primer pela neutralização de cargas negativas ↑ [K+ ] para fragmentos pequenos ↓ [K+ ] para fragmentos grandes
- 12. Importância dos dNTPs e íon magnésio na PCR • Deoxinucleotídeos trifosfato (dNTPs) • Associados ao DNA na forma de dNMPs • Sequestradores de Mg2+ • Mg2+ • Cofator da DNA polimerase • Especificidade e eficiência da enzima •Estabiliza ligação de dNTP/dNMP e primers à fita de DNA (↑ Tm) • Rendimento x Concentração crítica e empírica ↑ [Mg2+ ] ↑eficiência ↓especificidade
- 14. Função e relevância dos primers • Polinucleotídeos sintéticos de fita simples, de sequência complementar e flanqueadora à fita molde, com a extremidade 3´-OH livre
- 15. Thermus aquaticus DNA polimerase termorresistente • Antigamente: adição de enzima a cada novo ciclo • Taq DNA Polimerase • Dependente de cátions bivalentes (Mg2+ > Mn2+ >>>Ca2+ ) • Inibição por agentes quelantes (EDTA, citrato) Parque Nacional de Yellowstone / EUA
- 16. DNA molde • Qualidade • Cuidado com resíduos de processos de extração • SDS (dodecil sulfato de sódio) • Fenol • Corantes • Heparina/EDTA/Citrato • Polissacarídeos vegetais/carragenina • Poliestireno/polipropileno expostos à radiação UV • Quantidade • Excesso de DNA é prejudicial • Aumento de reação inespecífica
- 18. Parâmetros de termociclagem • Temperatura de anelamento ↑ Tanelamento ↑estringência ↓produtos inespecíficos ↓ Tanelamento ↑rendimento ↑ produtos inespecíficos • Tempo de extensão • DNA Polimerase: 35 – 100 nt por segundo a 72º C • Mínimo = 1 minuto • Excesso Atividade de exonuclase 5´-3´
- 19. Aditivos • Separação de fitas com alto conteúdo G:C • Estruturas secundárias fortes • Betaína (1 M) • DMSO (1-10%) • Formamida (1-10%) • Agentes estabilizantes da DNA polimerase • Redução da adesão de reagentes ao tubo • BSA (0,1 mg/mL) • Gelatina (0,1 – 1,0%) • Detergentes não-iônicos (<0,5%)
- 20. Inibidores • Derivados das próprias amostras ou do método de extração/purificação do ácido nucléico; • Fenol/álcoois • EDTA • Debris celular • Detergentes
- 23. Fonte: Koch WH, 2004. Nature Reviews Drug Discovery 3, 749-761 (Sep 2004)
- 24. Visualização dos resultados • Separação por eletroforese • Corantes/marcação fluorescente
- 25. Visualização dos resultados • Sequenciamento capilar • Dideoxinucleotídeos
- 26. Antes da PCR... • Clonagem!
- 27. Antes dos termocicladores... • Banho-maria!
- 29. Características de bons primers • Tamanho adequado • 15 a 25 pares de bases • Tamanho x Temperatura de dissociação • Temperatura adequada de dissociação (Tm) • Conteúdo G:C •Diferença entre primers < 5º C
- 30. Características de bons primers • Tamanho do fragmento a ser gerado • Tempo de extensão da polimerase •Especificidade Primers específicos x degenerados
- 31. Características de maus primers • Inter-complementariedade • Formação de dímeros (primer dimer) • Atenção à extremidade 3´
- 32. Características de maus primers • Auto-complementariedade • Formação de auto-dímeros e hairpins
- 34. Características de bons primers • Adequação para qPCR • Tamanho do amplicon •Junção exon-exon e contaminação com gDNA
- 35. Ferramentas online de suporte • Primer-BLAST www.ncbi.nlm.nih.gov/tools/primer-blast/ • Primer3 bioinfo.ut.ee/primer3-0.4.0/primer3 • IDT Oligo Analyzer 3.1 www.idtdna.com/calc/analyzer • UCSC PCR in silico genome.ucsc.edu/cgi-bin/hgPcr?command=start • RT Primer DB (específico para qPCR) • medgen.ugent.be/rtprimerdb
- 36. Problema detectado Workflow para desenho de primers Verificar • Temp. de anelamento • Temp. de dissociação • Tamanho do amplicon • Estruturas secundárias • Complementaridade • EspecificidadeNãoSim PCR para confirmação de funcionamento PCR para confirmação de funcionamento GenBank/NCBI Encontrar sequência da região desejada Encontrar sequência da região desejada PrimerBLAST Primer3, etc Desenhar primersDesenhar primers Registrar e arquivar resultados obtidos Registrar e arquivar resultados obtidos Reação adequada OK?OK? CHECAR PRIMERS!CHECAR PRIMERS!
- 38. PCR quantitativa • Nomenclatura • qPCR • qRT-PCR • Real-time PCR
- 39. Ensaios químicos disponíveis • SYBR Green I • Ensaio para 5´-nuclease (TaqMan) • Molecular Beacons • LightCycler • LUX Detecção de fluorescência! ↑ sensibilidade
- 40. SYBR Green I • Corante específico para DNA de fita dupla • Flourescência aumenta ao longo da reação • Não tem ação inibitória • Detecta produtos inespecíficos SYBR Green I Brometo de etídio
- 41. Ensaio para 5´-nuclase – Sondas TaqMan • Atividade 5´-nucleásica da Taq DNA Polimerase • Altamente específica, sem produtos espúrios • Duas marcações simultâneas são possíveis • Tecnologia baseada em FRET • Flourescence Resonance Energy Transfer
- 42. FRET - Transferência de energia por ressonância fluorescente • Fenômeno quântico entre duas moléculas muito próximas (10 – 100 Å), com bloqueio da emissão de luz • Quencher/bloqueador
- 43. Ensaio para 5´-nuclase – Sondas TaqMan
- 45. Ensaio para 5´-nuclase – Sondas TaqMan • Discriminação alélica
- 46. Conceitos importantes • Fases exponencial, logarítmica e plateau • Threshold • Baseline • Cycle threshold (CT) Ciclos Flourescência (produtosdePCR) Plateau Linear Exponencial Threshold
- 47. Ciclos 0 1 2 3 4 5 6 7 8 9 10 Amostra A 3 6 12 24 48 96 192 384 768 1536 3072 Amostra B 15 30 60 120 240 480 960 1920 3840 7680 15360 ~7,05 ~9,38
- 48. ↑ CT ↓ Qtde. inicial↑ CT ↓ Qtde. inicial Quantidade inicial CT Amostra A 3 9,38 Amostra B 15 7,05
- 49. Conceitos importantes • Quantidade inicial de amostra • Relação CT x Quantidade inicial
- 50. Conceitos importantes • Eficiência 2n = cópias em n ciclos Eficiência teórica= 100%
- 51. Conceitos importantes • Eficiência Qtde. inicial de DNA 1 10 100 1000 10000 100000 1000000 10000000 100000000 CT 40 35 30 25 20 15 10 5 1
- 52. Log 10 da qtde. inicial 0 1 2 3 4 5 6 7 8 CT 40 35 30 25 20 15 10 5 1 Conceitos importantes • Eficiência
- 53. E = 10(-1/slope) Conceitos importantes • Eficiência Eficiência 100% = 2n a cada ciclo = log 2 x Para um aumento de 10x = log 2 10 = 3,32 Slope (inclinação)
- 54. Conceitos importantes • Curva-padrão e blank sample • Gene de referência/constitutivo/normalizador • Gene de interesse Controle Infectado GAPDH IFN-γ Relação GAPDH IFN-γ Relação Amostra 1 20 20 1 5 40 8,0 Amostra 2 30 40 1,33 5 20 4,0 Amostra 3 60 80 1,33 10 50 5,0 Amostra 4 50 60 1,2 15 80 5,33 Amostra 5 40 50 1,25 10 60 6,0 Média + EPM 1,22 + 0,06 Média + EPM 5,66 + 0,67
- 55. Conceitos importantes • Curva de dissociação (Curva de melting) • Indica a temperatura em que 50% dos primers estão anelados; • Ligação específica do corante à fita dupla
- 56. Conceitos importantes • Curva de dissociação (Curva de melting) • Diretamente relacionada ao conteúdo G:C • Pode apontar amplificação inespecífica e dímeros de primers
- 57. Conceitos importantes • Curva de dissociação (Curva de melting) • Importante para distinção de amplicons/primers em protocolos multiplex
- 58. Fatores que afetam a eficiência da qPCR • DNA degradado • Produtos inespecíficos • Tamanho do amplicon • Condições de termociclagem • Prática laboratorial inadequada • Reagentes contaminados • Primers mal desenhados • Diluições mal feitas • Falta de calibração e manutenção • Pipetagem errada!
- 59. Funções da PCR quantitativa • Quantificação absoluta • Curvas-padrão • Quantidade conhecida de “cópias por volume” • Semelhante a um ELISA • Exemplos: carga viral, 16S bacteriano • Avaliação relativa • Ausência de curva-padrão • Gene de referência para normalização • Apresentada em aumento/redução em relação a um controle experimental • Exemplo: expressão gênica
- 60. CÁLCULOS E ANÁLISES CÁLCULOS E ANÁLISES Verificar dispersão entre replicatas biológicas e técnicas Verificar dispersão entre replicatas biológicas e técnicas Checar controles positivos (curva-padrão) e negativos (blank) Checar controles positivos (curva-padrão) e negativos (blank) Conferir curvas de dissociação (Tm)Conferir curvas de dissociação (Tm) Verificar ajuste do thresholdVerificar ajuste do threshold Análise de resultados Avaliar as curvas de amplificaçãoAvaliar as curvas de amplificação
- 61. Cálculos em qPCR – Quantificação absoluta • Regressão linear simples • Método PfafflMétodo Pfaffl Curva de cópias (plasmídeos)Curva de cópias (plasmídeos)
- 62. Cálculos em qPCR – Quantificação relativa • Relação matemática entre a expressão dos genes de referência e os de interesse • Método LivakMétodo Livak 22--ΔΔΔΔCTCT
- 63. Cálculos em qPCR – Quantificação relativa • Avaliada a conformidade de todos os outros parâmetros, a única informação útil será o CT • Exemplo: Controle Infectado GAPDH IFN-γ GAPDH IFN-γ Amostra 1 15,1 32,5 14,9 29,4 Amostra 2 15,4 33,1 15,5 29,1 Amostra 3 16,0 32,8 15,1 28,8 Amostra 4 18,1 31,5 15,8 29,9 Amostra 5 15,7 32,0 15,5 28,9
- 64. GAPDH IFN-γ Amostra 1 15,1 32,5 17,4 1,08 0,47 Amostra 2 15,4 33,1 17,7 1,38 0,38 Amostra 3 16 32,8 16,8 0,48 0,72 Amostra 4 18,1 31,5 13,4 -2,92 7,57 Amostra 5 15,7 32 16,3 -0,02 1,01 GAPDH IFN-γ Amostra 1 14,9 29,4 14,5 - -1,82 3,53 Amostra 2 15,5 29,1 13,6 - -2,72 6,59 Amostra 3 15,1 28,8 13,7 - -2,62 6,15 Amostra 4 15,8 29,9 14,1 - -2,22 4,66 Amostra 5 15,5 28,9 13,4 - -2,92 7,57 Controle Infectado ΔCT ΔΔCT 2 -ΔΔCT ΔCT Média ΔCT 16,32 Média ΔCT ΔΔCT 2 -ΔΔCT
- 66. Extração e manuseio de ácidos nucléicos • RNA = extremamente lábil! • Armazenamento correto • Dosagem e normalização das amostras • A260/280 e A260/230 > 1,8 • Contaminação com DNA genômico Confecção de cDNA • Normalizar a mesma quantidade (p.e. 2 µg) de RNA para todas as amostras; • Todo o procedimento deve ser realizado no gelo; • Ciclos de congelamento/descongelamento;
- 67. Escolha do gene de referência • É necessário ter opções! • Ex.: 18S, GAPDH, RPL4, HPRT para camundongo • Diferentes estímulos/tecidos/tratamentos podem ter a expressão do gene constitutivo alterada; •Preparar um pool com amostras-controle • 6 diluições em triplicata • Diferença entre o CT mínimo e máximo < 1,0 •Algoritmos gratuitos na internet •NormFinder: macro para Excel
- 68. Antes da qPCR... • Northern Blotting (RNA) • Southern Blotting (DNA)
- 70. PCR digital (dPCR) Baseada na compartimentalização da amostra • Gotículas (droplets) em emulsão = microfluidos • Chips com nanopoços Individualização das moléculas! Distribuição de Poisson Aplicações • Quantificação absoluta sem curva-padrão • Expressão gênica • Detecção de alelos raros e/ou mutações • Discriminação de baixas diferenças para CNV
- 72. Fonte: Baker M, 2012. Nature Methods 9, 541 (Jun 2012)
- 77. O que é importante saber? • Teoria e suas diferenças entre as técnicas • Vantagens e desvantagens de cada uma • Como aplicar a PCR ao seu projeto • Aplicações gerais e específicas • Boas práticas laboratoriais
- 78. Lucas Secchim Ribeiro [email protected] Dpto. Microbiologia Laboratório de Interação Microrganismo-Hospedeiro Sala C4 191 – Ramal 2735
Notas do Editor
- #10: Principalmente PRESENÇA x AUSÊNCIA!!!!!!!
- #11: TODAS AS OBSERVAÇÕES SÃO VÁLIDAS PARA REAL TIME!!!!!!!
- #12: Concentração normal de K+ no tampão: 50 - 100 mM
- #13: BAIXA ESPECIFICIDADE LEVA À FORMAÇÃO DE PRODUTOS INESPECÍFICOS DICA: DESCONGELAR E VORTEXAR BASTANTE O MgCl2!!
- #14: Remoção dos fosfatos beta e gama dos dNTPs para associação à fita de DNA
- #16: Thermus aquaticus = termofílica Gram Negativa ENZIMAS ANTIGAS: KLENOW ou T4 DNA Pol TAQ = MEIA-VIDA DE 40 MINUTOS a 95º C AUMENTAR A QTDE DE ENZIMA NÃO MELHORA O RENDIMENTO!!! ERROS DE PIPETAGEM DEVIDO AO GLICEROL! PROOFREADING = BLUNT NON-PROOFREADING = OVERHANGING (A)
- #17: Quantidade ideal = &lt; 10 ng/µL!!!!!!!
- #20: Detergentes não-iônicos: Tween-20, NP-40, Triton X-100
- #23: SEQUENCIAMENTO/NGS
- #25: APENAS NO END-POINT!!!! Explicar o que é end-point!
- #30: The Tm is also affected by the salt concentration of the reaction solution, because DNA duplexes are more stable at higher cation concentrations.
- #37: AO CHECAR PRIMERS, A REDUNDÂNCIA É BEM-VINDA!
- #41: Vantagens: custo e aplicabilidade quase infinita Desvantagens: possibilidade de detecção de falso-positivos, produtos inespecíficos











![Importância do tampão e íon potássio na PCR
• pH 8,3 pH 7,2 a 72º C
• Tris/ Tris-Cl
•Função enzimática da DNA polimerase
• K+
• Reduz a repulsão entre DNA/DNA e DNA/primer
pela neutralização de cargas negativas
↑ [K+
] para fragmentos pequenos
↓ [K+
] para fragmentos grandes](https://image.slidesharecdn.com/pcrquantitativa-lucassecchimribeiropgmicroufmg2015-1-150407210842-conversion-gate01/85/PCR-Quantitativa-Lucas-Secchim-Ribeiro-PGMicro-UFMG-2015-1-11-320.jpg)
![Importância dos dNTPs e íon magnésio na PCR
• Deoxinucleotídeos trifosfato (dNTPs)
• Associados ao DNA na forma de dNMPs
• Sequestradores de Mg2+
• Mg2+
• Cofator da DNA polimerase
• Especificidade e eficiência da enzima
•Estabiliza ligação de dNTP/dNMP e primers à fita
de DNA (↑ Tm)
• Rendimento x Concentração crítica e empírica
↑ [Mg2+
] ↑eficiência ↓especificidade](https://image.slidesharecdn.com/pcrquantitativa-lucassecchimribeiropgmicroufmg2015-1-150407210842-conversion-gate01/85/PCR-Quantitativa-Lucas-Secchim-Ribeiro-PGMicro-UFMG-2015-1-12-320.jpg)